-
Charlotte BRAMA,
-
Matthieu SANTIN,
Publié le 05/12/2018
Kernix est heureux d’avoir remporté le data challenge dédié au cartilage thyroïdien organisé à l’occasion des Journées Francophones de Radiologie 2018 ! Ce challenge d’intelligence artificielle autour de l’imagerie médicale était organisé par la Société Française de Radiologie en partenariat avec le centre Gustave Roussy et Centrale Supélec, et avait pour objectif de mettre en concurrence des équipes pluri-disciplinaires à travers cinq sujets de détection d’anomalie sur des IRM.
Kernix a participé à ce challenge au sein de l’équipe SynovIA dont faisait également partie le professeur Alain Blum, chef du service imagerie Guilloz du CHU de Nancy. La collaboration avec des radiologues tout au long de ce projet nous a permis d’acquérir une compréhension approfondie des problématiques en jeu et de mettre en place la méthodologie la plus appropriée. Notre approche de Deep learning nous a permis de détecter avec succès les anomalies sur des IRMs de cartilage thyroïdien.Cette démarche sera récompensée par une publication en fast track début 2019 dans la revue DIAGNOSTIC AND INTERVENTIONAL IMAGING !
Les journées francophones de radiologie se sont déroulées durant le week-end du 12 au 14 octobre 2018. C’est un lieu de rencontre idéal pour les radiologues et les industriels de l’imagerie médicale pour venir échanger autour des besoins des praticiens et des dernières innovations. Cette année, les avancées de l’intelligence artificielle étaient dans tous les esprits. En effet, la profession fait face à une pénurie de radiologues, soumettant ces derniers à des cadences toujours plus élevées tandis que les patients peuvent voir la qualité du service se dégrader. Les applications de l’intelligence artificielle pourrait permettre d’éviter au radiologue les tâches redondantes et chronophages, comme le traitement des informations du patient.
Les radiologues attendent ainsi de l’IA un gain d’efficacité afin de pouvoir se concentrer sur le coeur de leur métier, tout en réduisant les erreurs de diagnostic dû au manque de temps. C’est dans ce contexte qu’a été organisé le challenge IA de ces JFR : comment améliorer le diagnostic sur des images médicales en utilisant le deep learning ? Pour répondre à cette question, cinq thématiques ont été proposées : le cortex rénal, les fissures du ménisque, la radiographie mammaire, le cartilage thyroïdien et les lésions hépatiques. Dans chaque cas, il s’agit de repérer ou estimer une lésion ou une anomalie à partir d’images de scanner ou IRM. Les équipes participantes avaient donc pour objectif de proposer des algorithmes pouvant diagnostiquer des centaines, voire des milliers d’images. A terme, ce type de solution pourra être utilisé pour accompagner le radiologue dans un pré-diagnostic : dans le cas de la pathologie très courante de la fissure du ménisque, un algorithme donne un score à chaque image IRM pour refléter la probabilité de présence d’une fissure. Les résultats que nous avons obtenus dans cette pathologie ont une précision1 pour laisser entrevoir de telles applications.
Les organisateurs du challenge ont pu rassembler des jeux d’images sur chaque challenge auprès de différentes institutions de santé, malgré les difficultés dues au caractère sensible des données médicales. Une fois les données récoltées en nombre suffisant, les équipes se sont formées durant l’été, en mettant en relation des professionnels de l’IA avec des radiologues. Kernix s’est joint au service de radiologie du Dr Blum au CHRU de Nancy pour constituer l’équipe SynovIA.
Ce challenge regroupait des acteurs majeurs du domaine : IBM Watson, Facebook Research, Quantmetry… créant une émulation intéressante autour du challenge pour démontrer la puissance des outils issus du monde de l’IA et de la Data Science. Nous nous sommes ainsi reposés sur une démarche éprouvée chez nous : une compréhension des enjeux métiers par des échanges réguliers avec les radiologues, combinée à une expertise technique multidisciplinaire.
L’équipe du Dr Blum étant spécialisée dans les pathologies du ménisque, nous avons donc choisi naturellement de porter notre attention sur ce thème. L’objectif pour ce challenge est de prédire la présence d’une fissure sur une image IRM, de préciser sur quel ménisque elle se trouve et son orientation. Nous nous sommes aussi intéressé au challenge de la thyroïde en détectant une anomalie (classification binaire). Peu d’équipes l’ont abordé du fait de l’inhomogénéité des images : la forme de la thyroïde et de la trachée pouvait être sensiblement variable. Par ailleurs, il était même difficile pour les radiologues de prédire la présence d’une anomalie sur les thyroïdes. Mais ne serait-ce pas justement le plein intérêt d’un algorithme de deep-learning : affiner le diagnostic du médecin ?
Les échanges avec le Dr Blum et son équipe nous ont permis de comprendre et d’interpréter les images. Cette étape d’échange avec les métiers est indispensable à tout projet data en permettant la compréhension fine des contraintes du challenge.
Le challenge sur le ménisque comportait environ 1500 images d’IRM et celui sur la thyroïde en proposait le moins avec environ 500 IRM. Les images étaient globalement uniformes et centrées, ce qui facilitait leur traitement. En plus des images, nous avions un tableau avec les informations cibles (présence de fissure, sur quelle corne du ménisque, son orientation, la coupe de l’IRM).
Pour le ménisque, nous avons séparé les IRM en deux (parties haute et basse) puis réalisé un recentrage sur les cornes respectives (antérieure et postérieure) ainsi qu’un lissage de l’intensité. Un cropping selon des coordonnées fixes était suffisant étant donné l’uniformité des images et nous permettait de gagner du temps par rapport à une détection automatique de la zone du ménisque.
L’expertise des radiologues du CHU de Nancy nous a été très précieuse durant ce challenge afin de comprendre comment analyser les IRM et reconnaitres les anomalies, autrement dit quels motifs notre modèle allait devoir tenter d’identifier. Comme pour tous nos projets, nous avons pris soin de dédier le temps nécessaire à l’exploration des données au regard de la problématique. Voici un exemple du type d’images dont nous disposions :
Figure 1 : exemples d’IRM
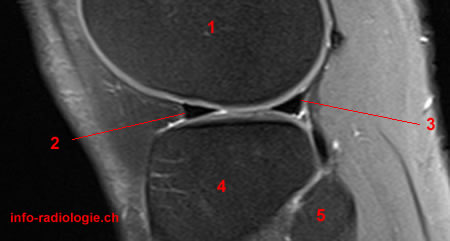 |
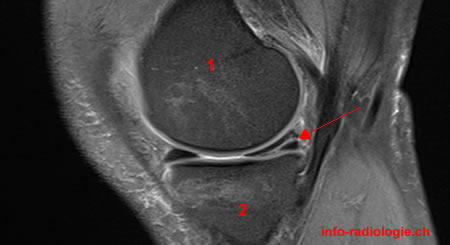 |
| IRM d’un ménisque sain | IRM d’un ménisque fissuré sur la corne postérieure |
Les IRM ci-dessus ont été réalisées selon une coupe dite « sagittale », comme illustré sur la coupe ci-dessous :
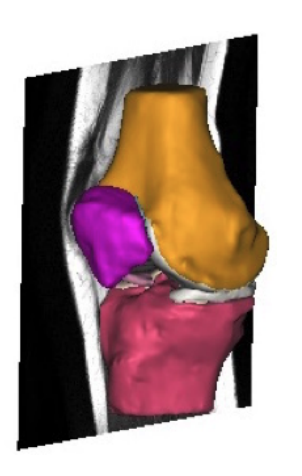
Figure 2 : Représentation de la coupe de l’IRM (Source : Automated Segmentation of the Meniscus – Felicia Aldrin)
Un ménisque sain est caractérisé par deux triangles dont la partie intérieure est sombre et homogène (voir figure 1 gauche), à l’inverse une fissure est caractérisée par une perturbation de l’homogénéité de cette zone à travers des zones blanches (voir figure 1 droite). La difficulté reposait dans la possible présence de kystes ou anomalies génétiques se manifestant de la même manière sans être des fissures.
Les traitements suivant ont été appliqués aux images d’IRM avant entraînement de l’algorithme (voir figure 3) :
Figure 3 : recentrage sur le ménisque
Le jeu de données a été séparé en échantillons d’entraînement (à partir duquel nous avons généré des images) et de test pour pouvoir évaluer les performances du modèle. Les images issues d’une même coupe étaient placées dans le même échantillon.
En ce qui concerne la thyroïde, voici un exemple des images dont nous disposions (environ 500 en tout) :
Figure 4 : exemples d’IRM de la thyroïde (Source : EURORAD)
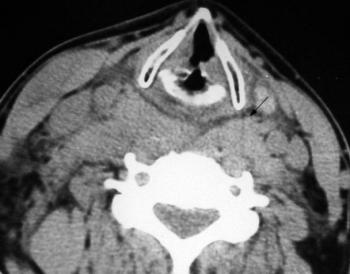 |
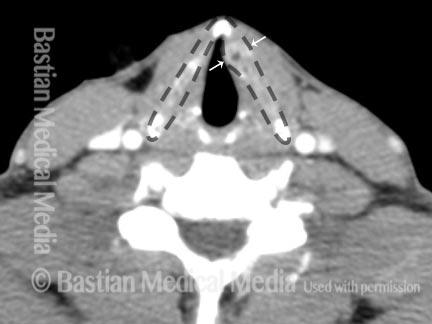 |
| IRM de la thyroïde (cartilage normal) | IRM de la thyroïde (cartilage anormal) |
Le problème de la thyroïde était particulièrement compliqué du fait du faible volume de données à disposition mais également de la difficulté du diagnostic qu’il est possible d’en déduire. En effet, les images étaient très disparates, ce qui est du à la variation de la forme du cartilage et la manière dont la coupe a été réalisée. La caractérisation de l’anomalie se fait donc différemment en fonction du type de coupe IRM. Des exemples de coupe sont illustrées dans la figure 6 ci-dessous :
Figure 5 :Illustration de différentes coupes d’IR
(Source :Radio-anatomie du larynx – M.Frikha, Y.Hentati)
En effet, les deux exemple présentés plus haut dans le figure 4 représentent le cas le plus simple ou un cartilage continu comme sur la droite est considéré comme normal et un cartilage discontinu comme sur la gauche sera considéré comme anormal. Cependant, nous pouvons également avoir le cas suivant où le cartilage sera diagnostiqué comme normal car la coupe n’est pas la même que pour l’exemple gauche de la figure 4 :
Figure 6 : IRM de la thyroïde (cartilage normal)
(Source : Radio-anatomie du larynx – M.Frikha, Y.Hentati)
Un cartilage anormal peut alors par exemple se caractériser par une coupure nette sans fermeture des différents morceaux, tandis que dans le cas d’un cartilage normal, même si la coupe entraîne une discontinuité, les différents morceaux de cartilage auront une fermeture apparente comme dans l’exemple ci-dessus (le diagnostic reste difficile à partir de ces images seules).
Nous avons également effectué un recentrage autour du cartilage à partir de coordonnées fixes, séparé le jeu de données en échantillons d’entraînement et de test et généré de nouvelles images comme pour le ménisque.
Figure 7 : Illustration du recentrage sur le cartilage
Nous avons considéré deux stratégies potentielles pour la modélisation : une basée sur l’extraction de zones d’intérêt au sein des images et une basée sur l’extraction de caractéristiques à l’aide de réseaux de neurones pré-entraînés.
Pour la première stratégie, nous avons utilisé la bibliothèque python OpenCV dédiée à l’analyse d’images et qui propose des extracteurs de points d’intérêt tel que présenté sur la figure 8 ci-dessous.
Figure 8 : Keypoints extraits avec OpenCV sur une image de ménisque
Ces points d’intérêt peuvent ensuite être exploités à l’aide de « descripteurs » (vecteurs) pour être utilisés comme variables à donner à un modèle. Nous avons testé d’extraire dix zones d’intérêt pour chaque IRM et de concaténer leurs descripteurs comme entrée d’un classifieur XGBoost mais les performances n’étaient pas convaincantes, notamment au niveau de la prédiction de l’orientation qui était un problème complexe.
En parallèle, nous avons essayé une approche de deep-learning basée sur du transfert learning. L’idée est d’exploiter les performances de réseaux de neurones profonds dédiés au traitement d’images déjà entraînés, sans pour autant repartir de zéro : nous n’avions ni la base d’images nécessaire, ni la puissance de calcul pour construire un réseau complet.
Pour cela, nous avons utilisé le modèle VGG162, développé par l’université d’Oxford et disponible dans la bibliothèque Python Keras. Il est entraîné à partir de la base ImageNet qui contient 1000 classes d’images différentes de tous types (objets, animaux, etc). Il existe d’autres réseaux, comme VGG19 et ResNet50 ayant des performances très intéressantes, mais dont l’architecture est plus complexe, pas forcément utile dans notre problème contenant peu d’images et impliquant des temps de calcul plus lourds.
Figure 9 : Schéma des couches du réseau VGG16. Pour ce challenge, nous avons modifié la dernière couche du réseau (entourée en rouge) pour obtenir une classification binaire.
Le réseau VGG16 est schématisé sur la figure 9. Il est composé de 5 blocs de convolutions et d’un bloc de neurones complètement connectés pour réaliser une classification en 1000 catégories. Notre objectif est de réutiliser l’architecture et les poids de ce réseau entraîné pour de la classification d’images, et de l’adapter à notre usage pour les images médicales. Pour cela, nous avons supprimé la couche finale et nous l’avons remplacé (en rouge sur le schéma) par le schéma suivant :
Pourquoi cette architecture ? Le choix s’est basé sur deux contraintes :
L’entraînement du modèle pour la prédiction de fissure s’est réalisé sur environ 3000 images de ménisque et a duré environ cinq heures, avec une machine dotée d’un processeur Intel Core i7 2,2 GHz et de 16 Go de RAM. Ce modèle a été réutilisé pour prédire l’orientation de la fissure et les anomalies de la thyroïde, en reprenant les poids préalablement calculés. Seule les 10 dernières couches ont été réentraînées. L’idée est de profiter d’un réseau ayant déjà « travaillé » des images d’IRM (celle du ménisque) qui sont plus proches des scanner de thyroïdes que les photographies d’ImageNet.
Pour évaluer les modèles réalisés par les différentes équipes, les organisateurs disposaient de jeux de données pour validation. Gare au surapprentissage !
Pour le challenge du ménisque, nous avons obtenu un score total de 0.84, classé 6ème sur les 13 équipes ayant participé. La meilleur équipe a obtenu un score de 0.91, bravo à elle !
En utilisant du deep-learning pour segmenter les zones autour du ménisques, ils ont pu avoir des images contenant uniquement les informations intéressantes. En outre, ils ont pu être très performants sur l’orientation de la fissure grâce à un traitement d’image préalable, qui faisait particulièrement ressortir le cas de fissures horizontales, plus facile à caractériser.
De notre côté, le modèle que nous avons proposé était très performant sur la détection de fissure et sur quel ménisque celle-ci se trouvait le cas échéant, avec un AUC de 0.97. Nous étions en revanche pénalisé sur l’orientation du ménisque, où nous n’avons pas pu dépasser 0.70 en score AUC.
Dans le cas du cartilage thyroïdien, nous avons obtenu un score AUC de 0.71 sur la détection d’anomalie. Si ce score qui peut paraître modeste en comparaison avec des scores obtenus pour la détection du ménisque, il est relativement honorable étant donné la complexité de simages. Même les radiologues de notre équipe était en difficulté pour diagnostiquer les images ! Ainsi, nous avons pu remporter ce challenge face à l’autre équipe ayant participé et ayant obtenu un score de 0.66.
La contrainte temporelle était aussi une des difficulté de ce challenge. En repartant des poids initiaux de VGG16 et en le ré-entraînant, nous arrivons à améliorer le score jusqu’à 0.85 sur un jeu de test. Nous espérons améliorer encore ce score en utilisant de la segmentation pour isoler uniquement les parties intéressantes de l’image, centrées autour de la thyroïde.
Cette première édition du challenge IA des JFR fut une expérience des plus enrichissantes ! Nous avons pu découvrir le secteur de l’imagerie médicale à travers une collaboration directe avec des experts du métier, et en apprendre plus sur leurs problématiques et comment l’IA peut améliorer leur productivité. Comme pour nos projets chez Kernix, nous avons ainsi pu apporter nos compétences en conception, analyse et machine learning pour répondre efficacement aux enjeux du challenge.
Les rencontres effectuées nous ouvrent de nombreuses perspectives et nous avons hâte de contribuer davantage au futur de la radiologie grâce à l’intelligence artificielle ! Encore merci à l’équipe organisatrice pour sa réactivité et son écoute tout au long du challenge qui s’est déroulé de manière fluide et agréable !